Profil d'intensité
Siril dispose d'un mode de profilage de l'intensité. L'utilisateur sélectionne une ligne entre deux points et Siril génère un graphique des valeurs des pixels entre ces deux points. Ce mode a plusieurs utilisations. Il peut être utilisé pour inspecter le profil d'intensité d'une étoile individuelle, il peut être utilisé pour profiler une galaxie entière, ou il peut être utilisé pour faire des spectrogrammes si vous avez un filtre à réseau de diffraction tel que le Star Analyzer SA-100 ou un vrai spectrographe.
Profil d'intensité de base
Pour établir un profil d'intensité de base d'une étoile ou d'un autre objet, sélectionnez le bouton Profil dans la barre d'outils inférieure. Siril passe alors en mode profilage et ouvre une petite boîte de dialogue.
Vous pouvez maintenant cliquer sur l'image principale et la faire glisser pour définir les points de départ et d'arrivée de la ligne que vous souhaitez profiler. Si vous maintenez la touche Maj enfoncée pendant que vous faites glisser la ligne, celle-ci se déplacera horizontalement ou verticalement.
Astuce
Lorsque la ligne de profil est exactement horizontale ou exactement verticale, les valeurs exactes des pixels peuvent être utilisées directement à partir de l'image. Lorsque la ligne de profil n'est ni horizontale ni verticale, les points à tracer ne tombent pas exactement sur un pixel et des valeurs de pixels interpolées de manière bilinéaire sont donc utilisées.
Un titre personnalisé pour votre graphique peut être saisi dans le contrôle au bas de la boîte de dialogue.
Astuce
Lors du traitement d'une séquence, il est possible d'afficher le numéro de l'image et le total dans le titre personnalisé en ajoutant () à la fin du titre. Par exemple, en saisissant Spectre solaire () comme titre pour une séquence de 5 images, les titres Spectre solaire (1 / 5), Spectre solaire (2 / 5), etc. seront générés. Les parenthèses sont ignorées et supprimées lors du traitement d'une seule image.
Types de profils
Utilisez les cases d'option pour sélectionner le type de profil que vous souhaitez. (Cliquez sur les exemples d'images ci-dessous pour les voir en taille réelle.)
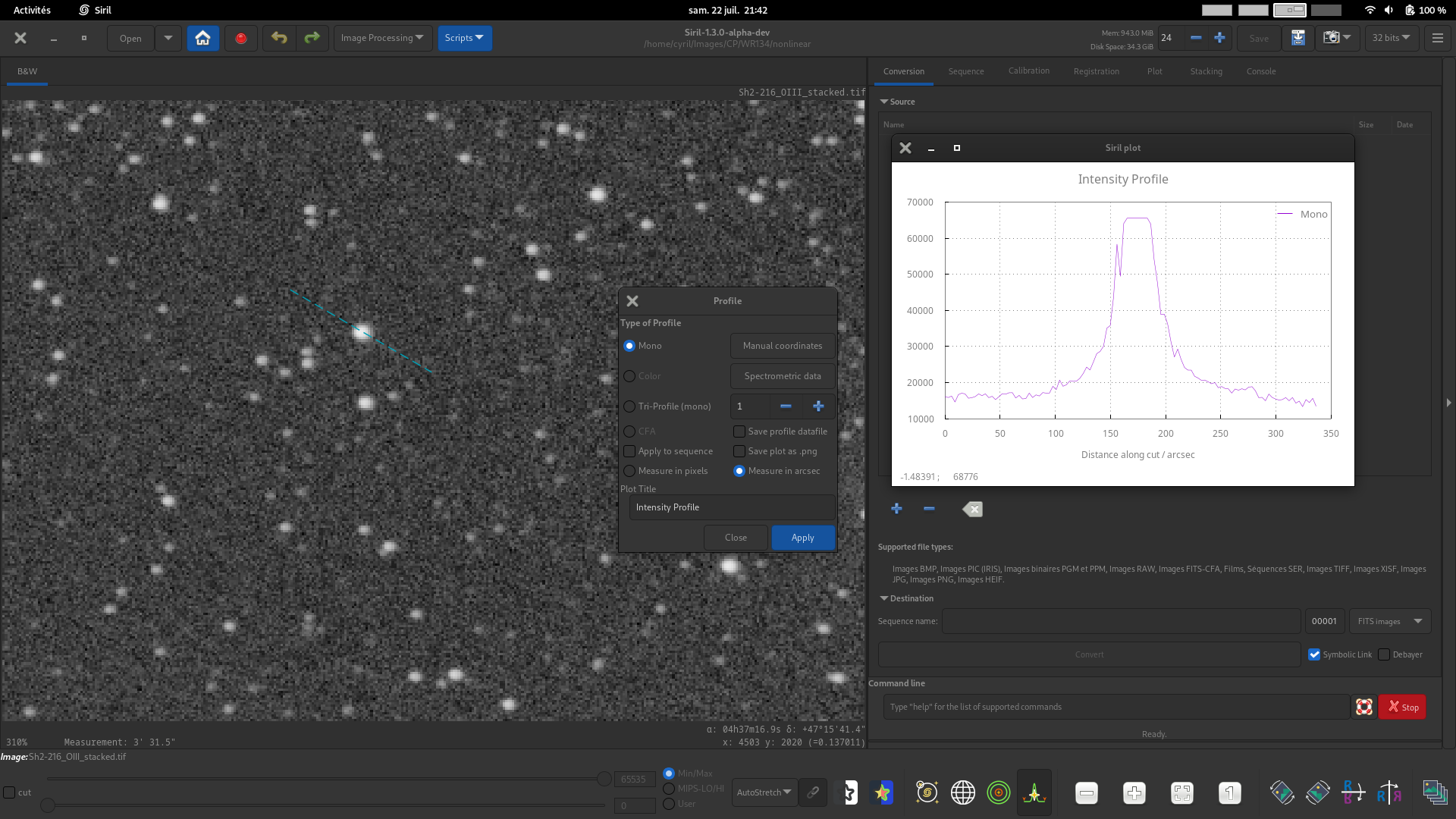
Profil mono. Pour les images mono ou couleur, générer un profil de luminance entre deux points. Ce mode peut être utilisé avec des données spectrométriques.
Astuce
Si une image couleur est chargée mais que le mode de profilage mono est sélectionné, le profil sera établi en fonction de la fenêtre d'affichage. Les fenêtres R, G et B fournissent des profils mono de leur canal respectif et la fenêtre RVB fournit un profil de luminance pondérant les 3 canaux de manière égale.
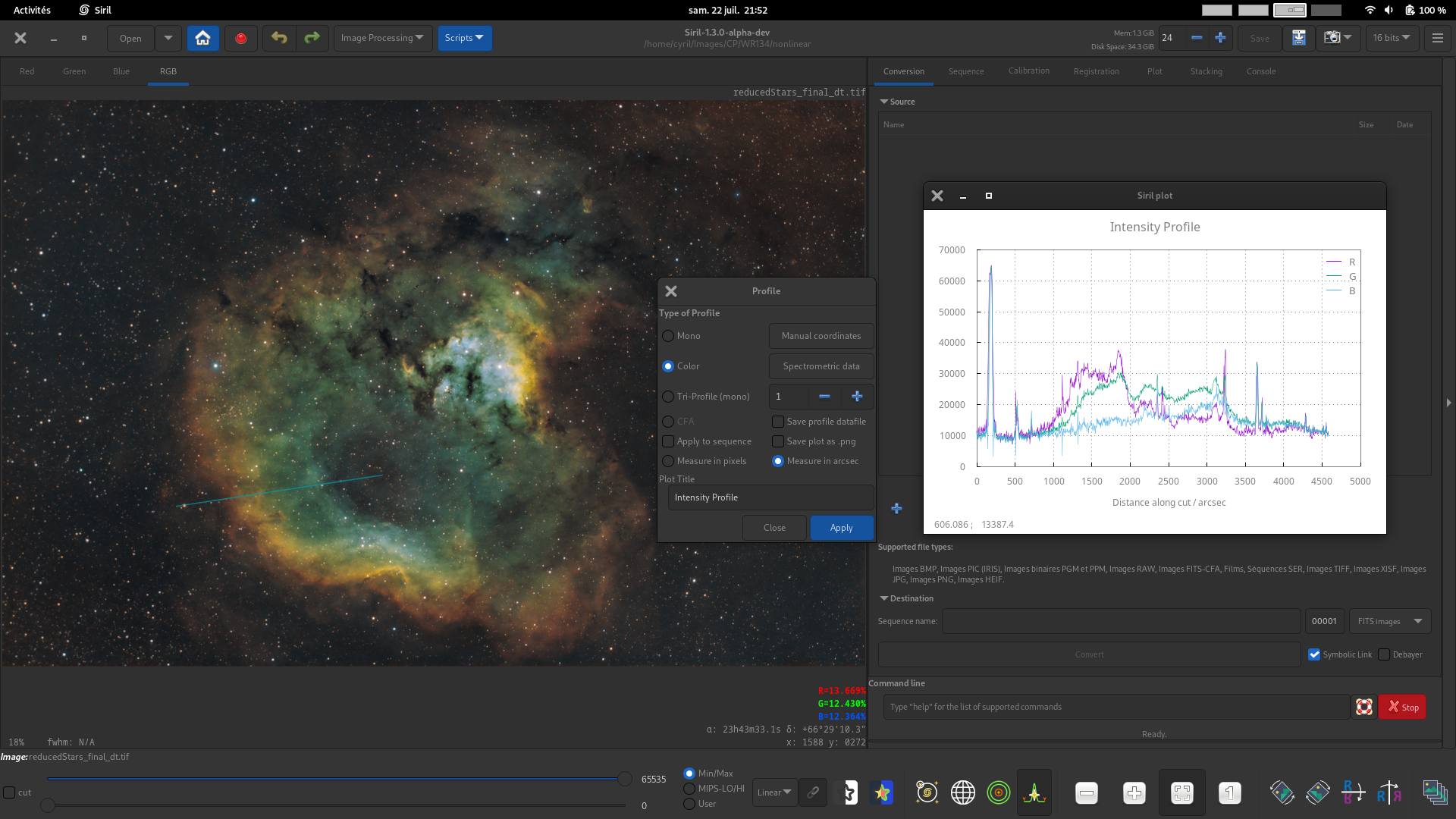
Profil de couleur. Pour les images couleur, générer trois profils pour les valeurs des pixels R, V et B entre deux points. Ce mode peut être utilisé avec des données spectrométriques.
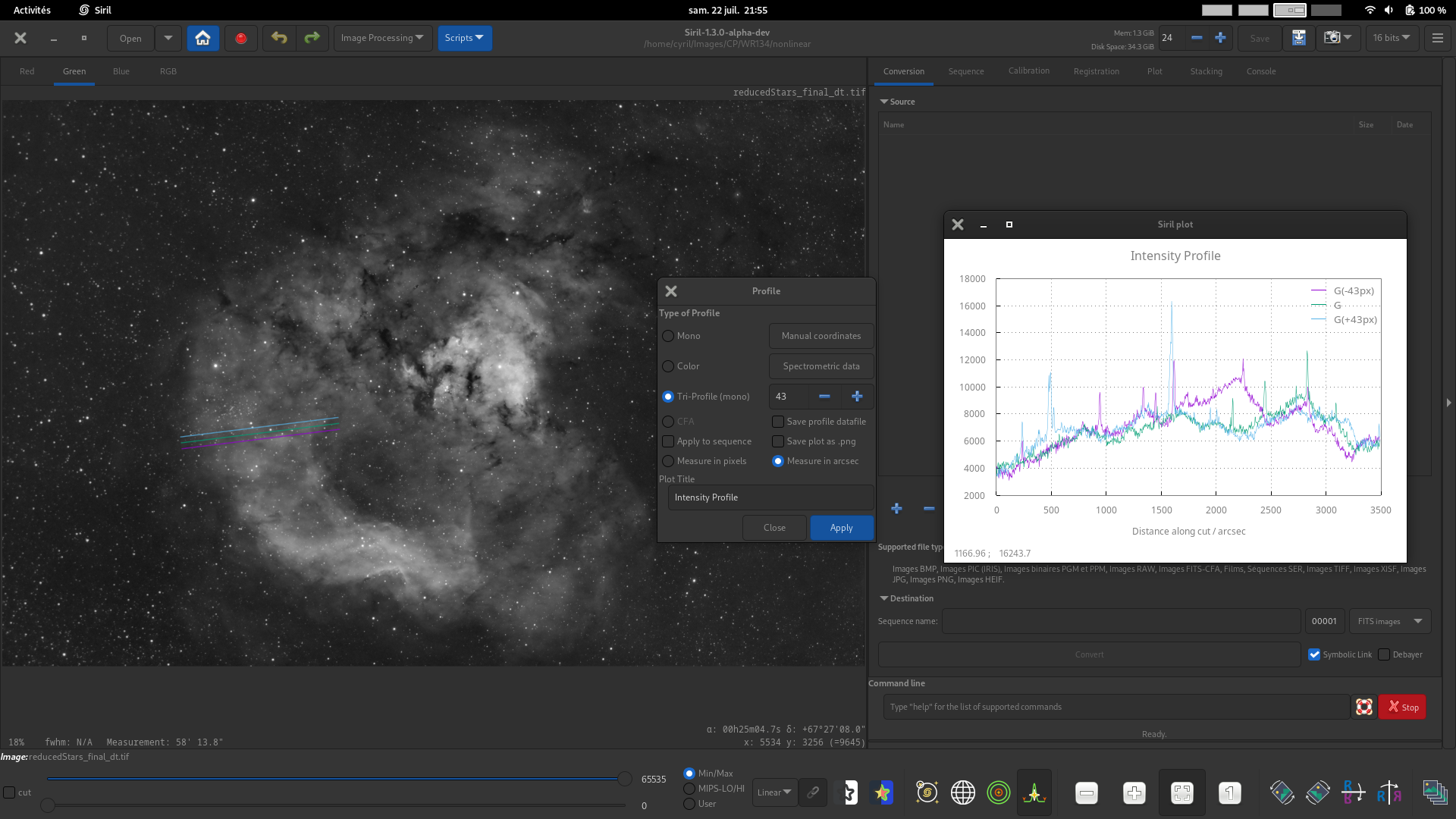
Tri-profil (mono). Pour les images mono ou couleur, génère trois profils de luminance parallèles et équidistants entre deux points. L'espacement entre les 3 profils peut être réglé à l'aide du bouton adéquate.
CFA. Pour les images comportant uniquement un motif de Bayer, générer quatre profils pour les quatre sous-canaux CFA entre deux points. Cela peut s'avérer particulièrement utile pour inspecter le profil des flats à motif de Bayer ou d'autres images à motif de Bayer avant qu'elles ne soient débayerisées.
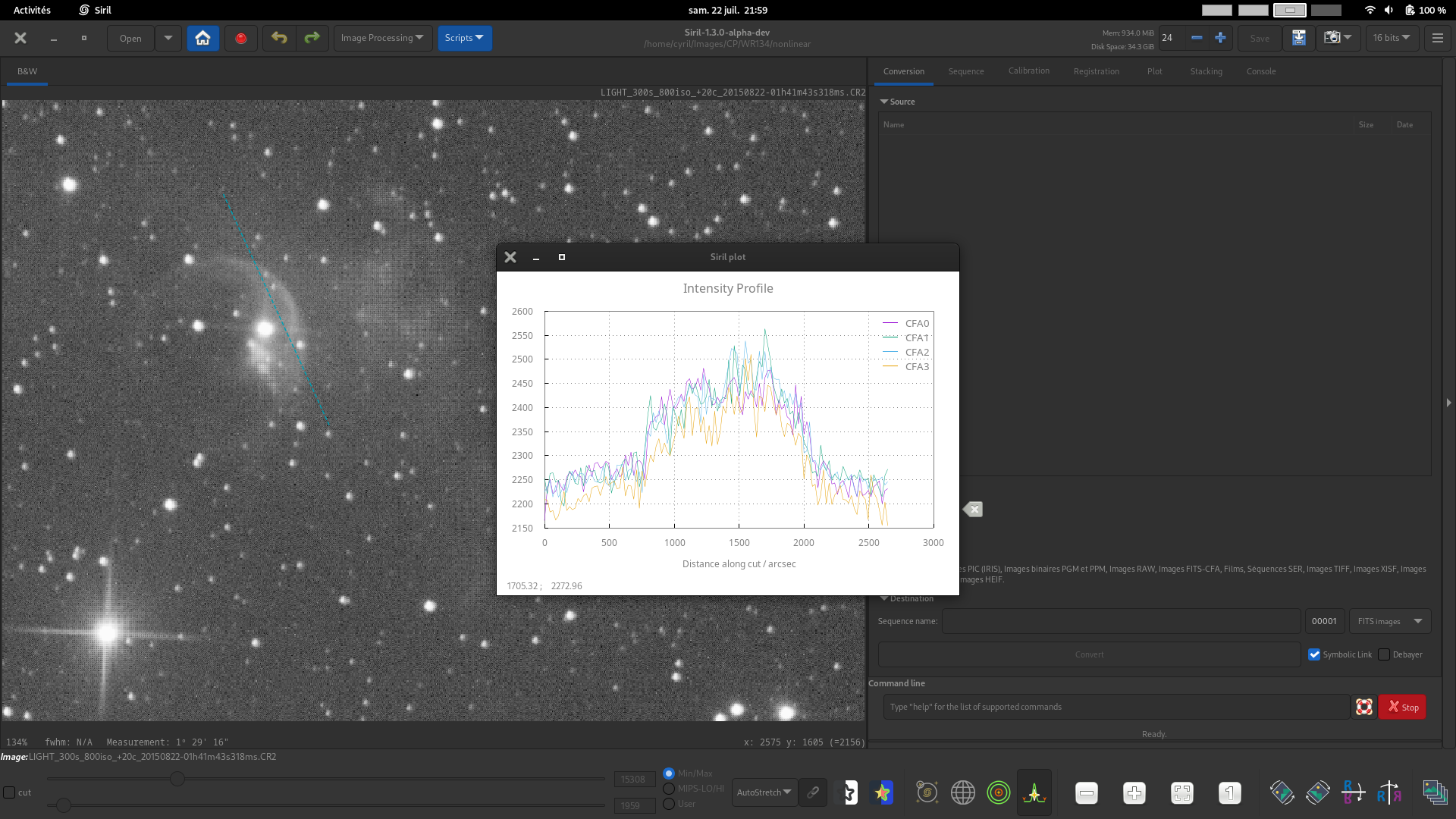
Cette image illustre l'utilisation du contrôle Titre personnalisé pour définir un titre personnalisé pour le graphe.
Note
Les options de données spectrométriques s'excluent mutuellement avec les modes de profilage tri-profil et CFA : le profilage tri-profil et CFA ignorera toute donnée de nombre d'onde fournie et l'option de largeur de profil.
Cliquer sur Appliquer pour générer le profil.
Saisie de coordonnées précises
Afin de faciliter la saisie des coordonnées de manière précise et répétée, une méthode de saisie manuelle est proposée. Cliquez sur le bouton Coordonnées manuelles et vous pourrez entrer les coordonnées X et Y des points de départ et d'arrivée de la ligne de profil. Si une ligne de profil est déjà tracée mais qu'un point n'est pas tout à fait à l'endroit voulu, vous pouvez utiliser cette boîte de dialogue pour affiner l'emplacement des points d'extrémité.
Si vous souhaitez définir un point final exactement à la position d'une étoile, faites une sélection rectangulaire autour de l'étoile et cliquez sur le bouton étoile correspondant à droite de la boîte de dialogue. Cette méthode est particulièrement utile pour la spectroscopie avec un filtre à réseau de diffraction, car elle permet de tracer le profil exactement au centre du spectre diffracté.
Astuce
Lorsque vous utilisez cette méthode pour la spectroscopie, l'étoile dont le spectre est représenté doit être sélectionnée comme point de départ et non comme point d'arrivée, sinon l'axe des nombres d'ondes sera inversé.
Note
Lorsque l'on utilise le mode CFA, les coordonnées sont données dans l'image d'entrée. Cependant, chaque canal CFA représente la moitié de la largeur et de la hauteur. L'axe des x dans le tracé du mode CFA est mesuré en pixels dans le sous-canal CFA, c'est-à-dire qu'il couvre la moitié du nombre de pixels qu'il couvre dans l'image d'entrée.
Mesures
La ligne de profil d'intensité peut être utilisée comme outil de mesure de deux manières :
En cochant la case Mesurer le profil, toutes les lignes de profil créées avec la souris seront mesurées, de manière similaire à la fonction de mesure rapide Ctrl-Maj-Glisser.
Dans la boîte de dialogue Coordonnées, il y a un bouton Mesurer. Il fournit la même fonction de mesure, mais vous permet de définir exactement les points d'extrémité, puis de mesurer la ligne de profil à la demande. En sélectionnant des étoiles, des planètes mineures ou des noyaux de comète comme points d'extrémité, comme décrit ci-dessus, les mesures entre deux corps célestes peuvent être effectuées très précisément (avec une précision inférieure au pixel).
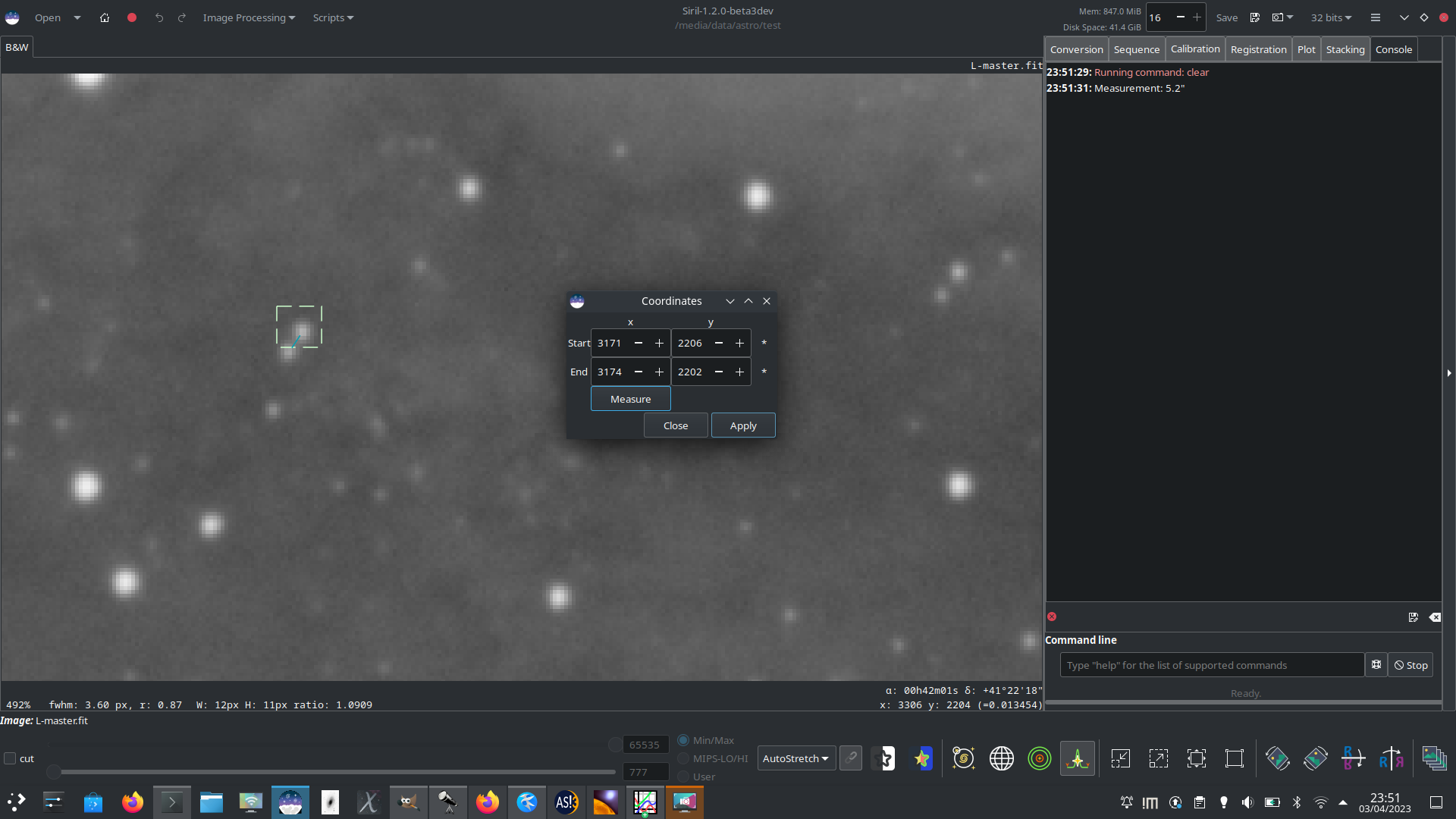
Ici, deux étoiles proches ont été sélectionnées et placées comme points d'extrémité, et la séparation entre elles a été mesurée à 5,2 arcsec. Cette méthode pourrait être utilisée pour étudier les binaires proches ou pour trianguler la position d'une planète mineure.
Note
La fonction de mesure de Siril fait l'approximation du petit angle pour la séparation angulaire \(\theta\). Le terme d'erreur le plus significatif est proportionnel à \(\theta^3\) et est inférieur à 1% pour les mesures jusqu'à 10° : il est donc valide pour la plupart des utilisations astrométriques, mais deviendra imprécis pour les grandes mesures sur des images à très grand champ. Un avertissement sera écrit dans les logs pour les mesures supérieures à 10°.
Spectroscopie
Vous disposez peut-être d'un spectrographe ou d'un filtre à réseau de diffraction. Dans ce cas, vous pouvez utiliser les fonctions supplémentaires du mode profilage pour produire un spectre. Cliquez sur le bouton Données spectrométriques pour saisir les données relatives aux mesures spectrales.
Afin de réduire le bruit, une largeur de coupe peut être spécifiée. Si cette valeur est supérieure à 1, une valeur moyenne de plusieurs pixels perpendiculaires à la ligne de coupe sera utilisée à chaque point au lieu d'une valeur unique. Une largeur plus importante réduira davantage le bruit, mais si l'on utilise un réseau de diffraction, le spectre d'une étoile est assez étroit, de sorte que la largeur du profil est limitée avant de sortir de la région de l'image où se trouvent les données spectrales.
Pour calibrer l'axe des nombres d'ondes, il est possible de sélectionner deux points sur le graphique avec des nombres d'ondes ou des longueurs d'onde connus, par exemple des raies d'absorption connues. Pour ce faire, cliquez d'abord sur le bouton Sélectionner point 1, et cliquez sur l'image à l'endroit correspondant au premier nombre d'ondes connu. Les coordonnées x et y choisies s'accrocheront au point le plus proche de la ligne de coupe. Si vous cliquez au mauvais endroit, il vous suffit de cliquer à nouveau sur le bouton et de resélectionner le point. Répétez ce processus pour le deuxième nombre d'ondes ou longueur d'onde connu, en utilisant cette fois le bouton Sélectionner point 2.
Vous pouvez également choisir de tracer votre spectre avec un axe x montrant la longueur d'onde (par défaut) ou le nombre d'ondes à l'aide de la liste déroulante.
Note
L'étalonnage de l'axe des abscisses repose sur l'approximation du petit axe, c'est-à-dire que la distance le long de l'axe x est proportionnelle à la longueur d'onde. Cela provient de l'équation de diffraction \(d \sin(\theta) = m \lambda\). Cela donne la distance diffractée le long d'un axe x perpendiculaire au réseau \(x = D \tan\left(\arcsin\left(\frac{m \lambda}{d}\right)\right)\) et l'erreur sur l'axe x est inférieure à 1% tant que l'angle diffracté est inférieur à 9 degrés. Ceci est vrai pour de nombreux réseaux de diffraction, par exemple le Star Analyser SA-100 a un espacement de 100 lignes / mm. Pour les émissions d'hydrogène alpha d'une longueur d'onde de 656nm, cela donne un angle de diffraction de premier ordre de 0,125 degrés.
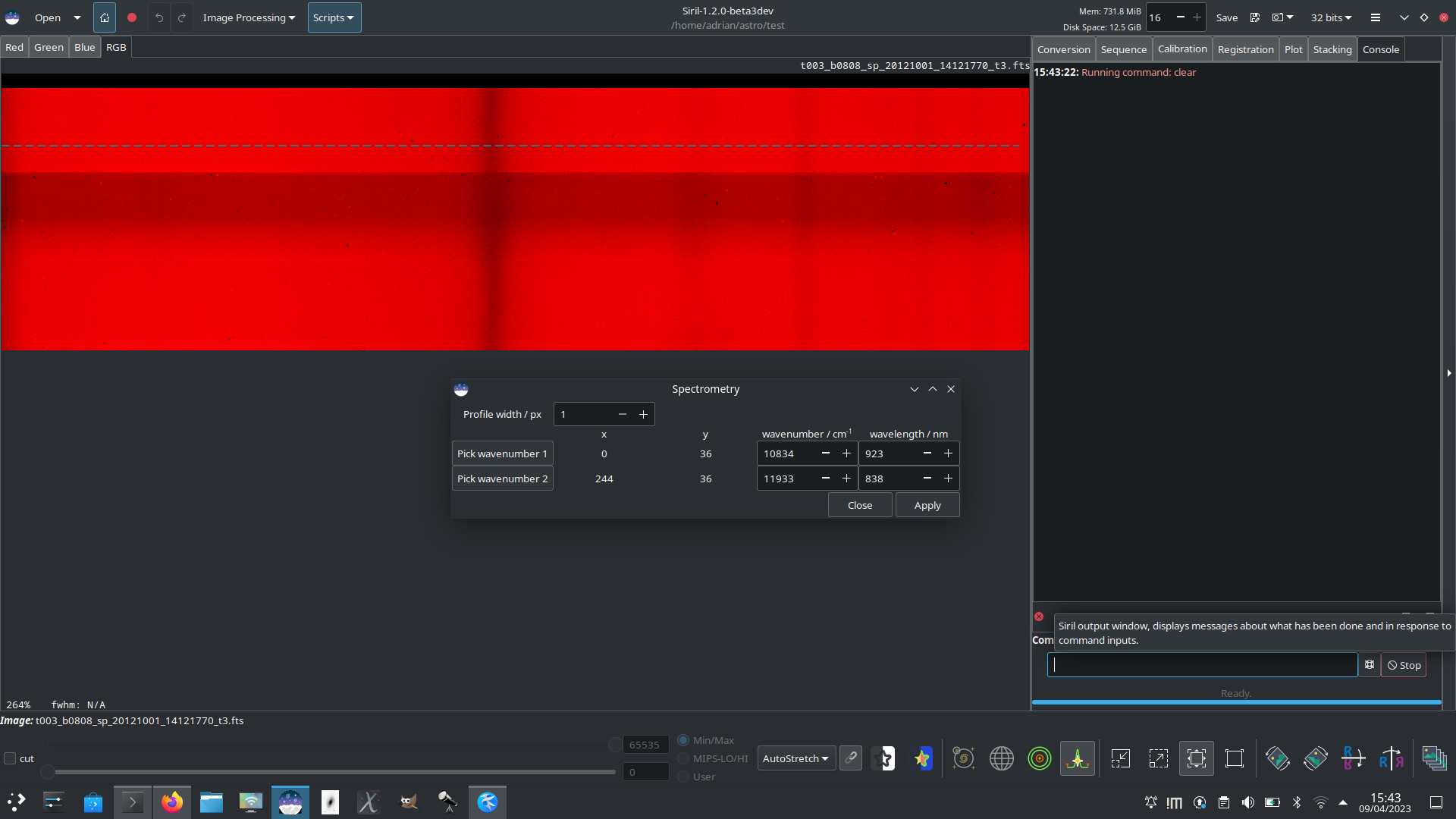
Une fois que vous êtes satisfait de vos choix, cliquez sur Appliquer pour revenir à la boîte de dialogue principale de profilage. Vous pouvez maintenant cliquer sur Appliquer pour générer votre spectre.
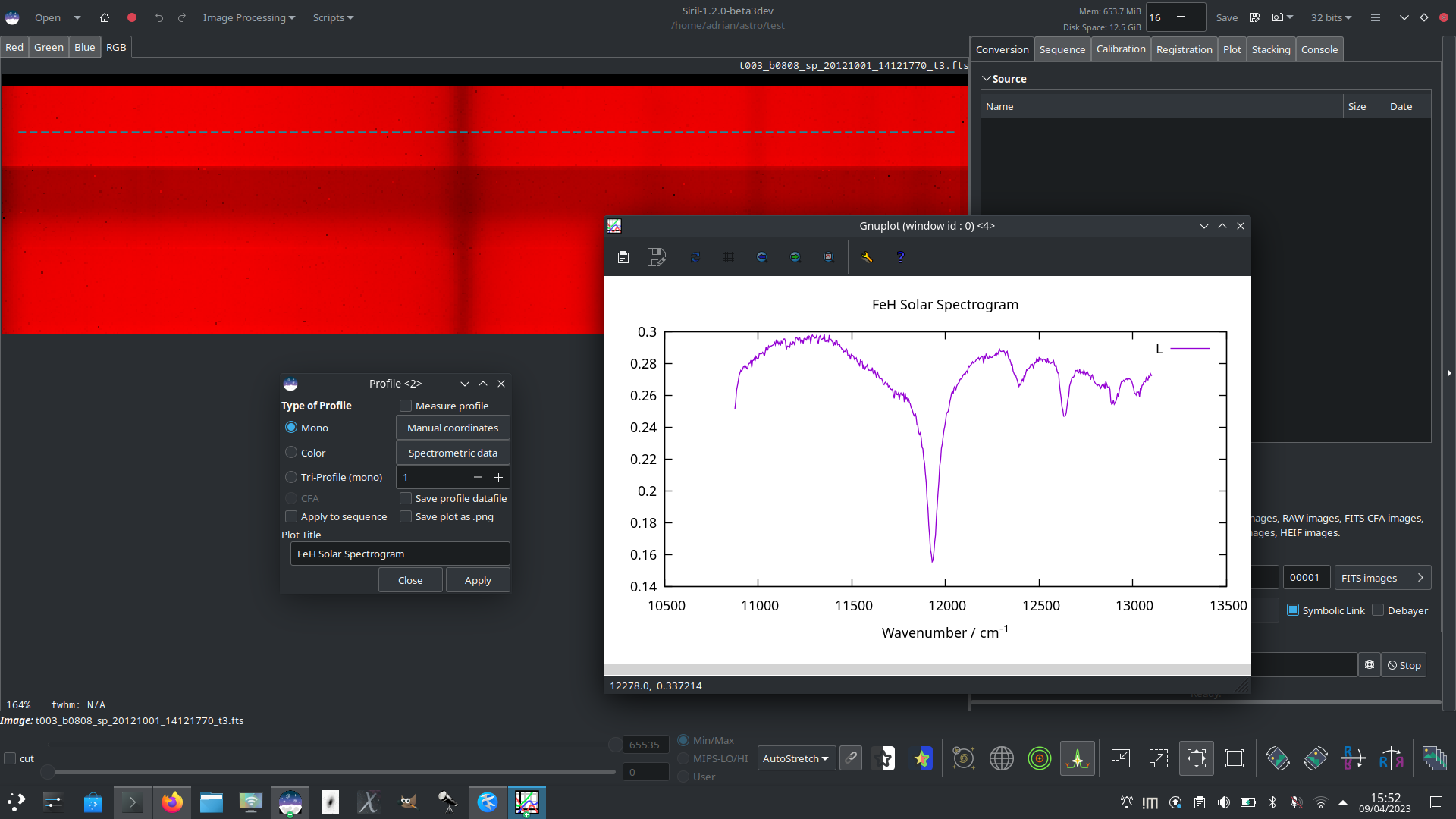
Voici un spectre solaire de FeH capturé à l'aide du télescope THEMIS, moyenné sur 15 pixels par colonne pour améliorer le rapport signal/bruit et avec l'axe des nombres d'ondes étiqueté à l'aide de la fenêtre contextuelle des données spectrométriques.
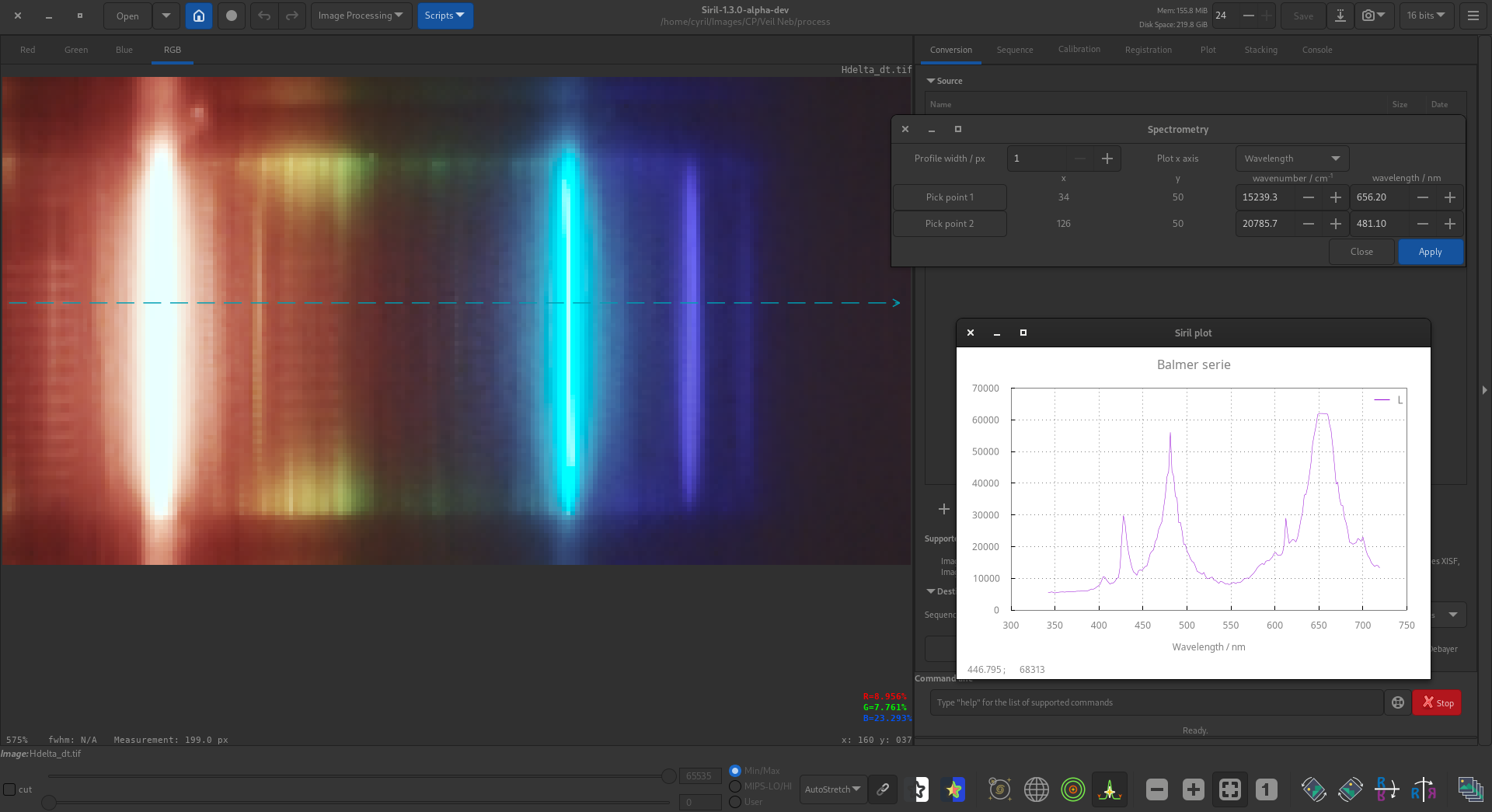
À titre d'exemple, voici les raies de l'hydrogène de la série Balmer prises à travers un réseau de diffraction. Deux raies ont été utilisées (\(text{H}_\alpha\) et :math:text{H}_beta`) pour calibrer le spectre.
Note
Le nombre d'ondes, tel qu'il est utilisé en spectroscopie et dans la plupart des domaines de la chimie, est défini comme le nombre de longueurs d'onde par unité de distance, généralement en centimètres (cm \(^{-1}\)) :
où lambda est la longueur d'onde. Il est parfois appelé nombre d'ondes spectroscopiques. Il est égal à la fréquence spatiale.
Outil graphique de Siril
La fonction de profilage utilise l'outil graphique interne de Siril pour afficher les différents profils. Avec les fichiers *.dat produits, vous pouvez toujours utiliser l'outil graphique de votre choix pour explorer les données sous-jacentes.
Un clic droit n'importe où dans la surface du graphique fera apparaître un menu contextuel pour :
Afficher/masquer la grille et la légende
Exporter la vue actuelle dans le presse-papiers,
*.pngou*.svgSauvegarde des données sous-jacentes dans un fichier
*.dat
Menu contextuel du graphe de Siril
Notez que toutes les exportations tiennent compte du zoom/pan actuel, tandis que l'enregistrement dans un fichier dat exporte des données non filtrées.
Les interactions suivantes de l'interface graphique sont disponibles :
Cliquer + glisser pour dessiner une sélection. Le zoom est réglé sur la zone sélectionnée lorsque la souris est relâchée.
Cliquer + glisser pour effectuer un panoramique de la vue courante.
Ctrl + Défilement pour zoomer/dézoomer.
Double-clic pour rétablir la position/le zoom par défaut.
Commandes
Ligne de commande Siril
profile -from=x,y -to=x,y [-tri] [-cfa] [-arcsec] { [-savedat] | [-filename=] } [-layer=] [-width=] [-spacing=] [ {-xaxis=wavelength | -xaxis=wavenumber } ] [ {-wavenumber1= | -wavelength1=} -wn1at=x,y {-wavenumber2= | -wavelength2=} -wn2at=x,y] ["-title=My Plot"]
Ligne de commande Siril
seqprofile sequence -from=x,y -to=x,y [-tri] [-cfa] [-arcsec] [-savedat] [-layer=] [-width=] [-spacing=] [ {-xaxis=wavelength | -xaxis=wavenumber } ] [{-wavenumber1= | -wavelength1=} -wn1at=x,y {-wavenumber2= | -wavelength2=} -wn2at=x,y] ["-title=My Plot"]